肠道菌群提取细菌DNA的方法
Release Time:2020-08-22肠道菌群提取细菌DNA实验目的:
肠道菌群提取细菌DNA实验是建立一种简便的从肠道菌群中提取基因组DNA的方法,用于肠道细菌重复基因间共有(ERIC)-PCR检测。
肠道菌群提取细菌DNA实验方法:
比较了Tr??is-EDTA缓冲液,chelex-100,超纯水,2%十二烷基硫酸钠和10%Triton-100(经超声处理和不经超声处理)提取细菌DNA的方法,并与商业粪便DNA提取试剂盒方法进行了比较。被认为是DNA提取的金标准。比较是基于ERIC-PCR反映的DNA的产量和纯度以及微生物的结构和性能指标。
肠道菌群提取细菌DNA实验结果:
通过chelex100的方法获得的DNA的产量和纯度与通过粪便DNA试剂盒获得的DNA相似。通过chelex100方法提取的DNA与通过粪便DNA试剂盒提取的DNA获得的ERIC-PCR结果基本相同。
肠道菌群提取细菌DNA实验结论:
鉴于其简单性和成本效益,建议将chelex方法用于ERIC-PCR实验。它适用于从肠道微生物中提取总DNA,特别适用于处理大量样品。
肠道菌群提取细菌DNA实验介绍
※ 动物的肠道内有大量,活跃和复杂的微生物群落[1]。至少有400-500种不同的微生物,构成一个复杂的生态系统。
※ 它们在定植抗性中发挥着重要作用,即在胃肠道中防止病原体定植和自发机会性微生物的生长。合成或代谢潜在致癌物并产生抗致瘤产物的肠道细菌可能与结直肠癌有关,这是美国第二大最常见的癌症死亡原因[4]。
※ 益生菌被定义为一种生物,在摄入一定数量后,其健康益处超出宿主固有的基本营养[5,6]。在人类中,益生菌可有效预防眼袋炎的发作和复发[7,8],在小鼠中,它们可有效预防实验性结肠炎并减少肠道细菌的移位[9,10]。
※ 共生肠道菌群已通过基于培养的技术和分子检测进行了监测[11,12]。肠细菌重复基因间共有(ERIC)-PCR使用靶向短重复序列的寡核苷酸,这些短重复序列分散在各个细菌基因组中[11]。共生肠道菌群可以根据扩增产物的电泳图谱在属,种和菌株水平上鉴定[13-15]。
※ 这些测定法监测动物肠道细菌的能力取决于PCR的效率。从肠道菌群获得PCR指纹图谱的关键因素之一是DNA提取程序的效率。
※ 通常,在提取过程中必须特别考虑两个因素。首先是使DNA产量最大化。第二是确保提取的DNA可以进行多种酶促处理,例如PCR扩增[16]。换句话说,最大的挑战是从肠道菌群中提取高质量的PCR兼容DNA。已经评估了几种使用去污剂,蛋白水解酶,溶菌酶,机械破坏,单独或以多种组合的温度变化,DNA粪便迷你试剂盒等方※ 法进行细菌细胞壁裂解和DNA提取的方法。尽管DNA粪便迷你试剂盒方法很方便,快速,高效,由于成本高而未得到广泛应用[17-20]。
※ 在这项研究中,将5种提取细菌DNA的方法与使用粪便DNA试剂盒(FDK)的方法进行了比较。这项研究的目的是建立一种经济,简单,方便的方法,从肠道菌群中提取基因组DNA,以进行ERIC-PCR检测。
肠道菌群提取细菌DNA所需的材料和方法
※ 粪便样本的收集和准备:
从20个10天大的健康幼鹅中收集新鲜的粪便样本。没有受试者接受抗生素,益生菌或益生元。遵照疾病控制与预防中心提供的《机构动物护理和使用指南》进行小鹅护理和实验程序。随机选择两只幼鹅,并分别从直肠收集粪便。将样品收集在无菌袋中,冷冻等级,并立即送至实验室。
总DNA提取
六种提取方法均重复3次。首先将每个样品(20 mg)解冻并悬浮在5.0 mL磷酸盐缓冲盐水(PBS; pH 7.2)中。为了除去粪便颗粒,在4℃下以100g离心15分钟。将得到的上清液在4 000°C下以13000 g离心10分钟。然后通过将其悬浮在1.5mL丙酮中洗涤3次。为了除去粪便中潜在的PCR抑制剂,每种制剂在4℃下以13 000 g离心10分钟[21]。弃去上清液,并按以下步骤处理沉淀物。
(1)TE煮沸提取DNA(T method):
它是对Li等人[22]描述的细菌DNA提取方法的改进。将沉淀物悬浮在200μLTE缓冲液[10 mmol / L Tris-HCl(pH 8.0),1 mmol / L EDTA] [23]中,并将混合物在涡旋混合器上短暂混合。将悬浮液置于沸水浴中1分钟,进行3次冻融循环,在-70℃下3分钟和100℃下2分钟交替,然后以10000g离心5分钟。将上清液的100μL等分试样转移到无菌试管中,并保存在-20°C下直至进行PCR测试。
(2)超纯水法提取DNA(UW法):
将200μL的超纯水等分试样加入到沉淀物中,并如上文针对TE缓冲液所述处理悬浮液。
(3)chelex100方法提取DNA(C方法):该方法是Emi Suenaga [24]描述的细菌DNA提取方案的改进。将200μLChelex-100(5%)和0.2 mg蛋白酶K的等分试样加入沉淀中,并将样品在56°C的水浴中温育30分钟。然后将混合物在涡旋混合器上短暂混合,并以10000 g离心5分钟。将上清液的100μL等分试样转移到无菌试管中,并保存在-20°C下直至进行PCR测试。
(4)SDS方法提取DNA:除将200μL非离子型洗涤剂混合物(2%SDS含10%Triton X-100)代替200μLTE缓冲液外,按上述方法处理TE缓冲液[18]。
(5)SDSS法提取DNA:将200μL等分试样的含有10%Triton X-100的2%SDS加到沉淀中,并在涡旋混合器上短暂搅拌混合物。将悬浮液超声处理15分钟,然后按上述关于TE缓冲液的方法处理[18]。
(6)FDK方法提取DNA:使用Fecal DNA Kit™(中国田里市)处理沉淀,并根据制造商的说明使用旋转柱纯化DNA。
DNA浓度和纯度的测量
用分光光度法(BIO-RAD Smart Spec 3000;美国)测定DNA的浓度和纯度。为此,在260 nm下测量DNA吸光度(μgDNA / g样品; 1 A 260 = 50μg/ mL DNA),并在280 nm下检查蛋白质杂质[25]。通过Windows的Excel 2003对每种DNA提取方法的浓度和纯度进行统计分析。
通过不同提取方法检测基因组DNA
对于每种测试方法,使用4%的含溴化乙锭的0.5%琼脂糖凝胶分析从一式三份样品中提取的基因组DNA的存在和质量。将通过每种方法提取的10微升DNA添加到凝胶中,并在150 V下电泳30分钟。使用凝胶成像系统(Bio-Rad)以标记图像文件格式(TIFF)文件获取凝胶图像。除非另有说明,否则使用从1或2个一式三份样品中提取的基因组DNA进行以下分子程序。
ERIC-PCR和统计分析
为了对粪便样品中的细菌种群进行指纹识别,将粪便总DNA用作ERIC-PCR的模板,ERIC引物的序列为E1(ERIC1R):5'-ATGTAAGCTCCTGGGGATTCAC-3'和E2(ERIC2):5 Versalovic等[11]描述了“ -AAGTAAGTGACTGGGGTGAGCG-3”。 ERIC-PCR在Di Giovanni等[26]描述的条件下进行。
如Melanie W Syrmis [27]所述,我们通过琼脂糖凝胶电泳分析了PCR产物。我们使用凝胶成像系统(Bio-Rad)将凝胶图像作为TIFF文件获取。
为了解释ERIC-PCR指纹图谱,使用了具有成对相似系数(Cs)的Quantity One软件包(Applied Bio-Rad)[28]。当观察到任何谱带差异时,会将指纹分配给其他类型。谱带强度的变化不视为谱带差异。当Cs大于90%时,指纹被认为是相似的;当Cs为0时,指纹被认为是不同的;当Cs为100%时,指纹被认为是相同的[28]。
实验结果
通过不同方法获得的DNA的产量,纯度和质量
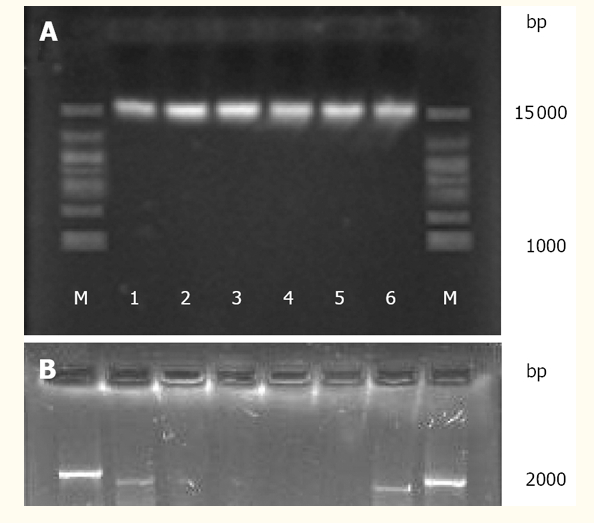
表六显示了从六种提取方法获得的基因组DNA的产量,纯度和质量。通过A260 / 280比值评估提取的DNA的质量,接近1.8的值表明DNA提取物质量好,几乎没有蛋白质污染[25]。尽管通过所有六种方法(图(图1A),1A)获得了高分子量DNA(约15 kb),但在不同方法之间观察到DNA的产量和纯度差异。例如,SDSS和SDS方法产生的DNA量最大(分别为1820.12μg/ g和1710.95μg/ g),但A260 / 280比率最低(分别为1.49和1.54),这意味着DNA纯度最低。 T和UW方法产生的DNA量最少(分别为601.52μg/ g和586.03μg/ g)和较低的DNA纯度(平均A260 / 280比为1.58和1.60)。 C和FDK方法产生的DNA量相似(分别为1301.52μg/ g和1326.07μg/ g)和相似的A260 / 280比例(分别为1.84和1.82)。通过两种方法获得的DNA的纯度最高。




 购物车
购物车 



